



PK(药代动力学)分析主要研究药物在体内的吸收、分布、代谢和排泄过程,其涉及的相关图表包括血药浓度-时间曲线、PK参数计算、剂量线性分析等。
R 是一款开源免费、扩展性强、脚本化操作的统计编程语言和环境,专为数据分析和可视化设计,适用于早期研究、探索性分析、内部决策支持等非递交阶段。正式递交仍需使用已验证的商业软件(如Phoenix WinNonlin、SAS),以确保合规性。
本文演示使用R完成PK分析全流程,包含:
➢
非房室模型(NCA):用于计算关键PK参数(如Cmax、AUC),无需假设房室结构,适用于早期药物研发阶段。
➢
Power Model分析:探索剂量与暴露量的线性关系,支持剂量优化决策。
➢
可视化分析:通过专业图表(如线性和半对数药时曲线图)直观呈现药物动力学特征。
➢
自动化报告生成:利用r2rtf包实现从数据到Word报告的一键输出,减少人工操作误差。
流程结合了开源工具(如PKNCA、ggplot2)与传统商业软件(WinNonlin、SAS)的对比验证,确保分析结果的可靠性。
# 核心分析包library(tidyverse)library(readxl)library(PKNCA) # 非房室分析library(r2rtf) # RTF输出# 辅助工具library(knitr) # 交互式表格
PART 01
数据模拟

原始数据模拟
pc <- read_xlsx(path=str_c(getwd(),"/outputs/pc.xlsx"))# 查看模拟数据knitr::kable(pc[1:10, 1:6])

PART 02
非房室分析(NCA)

关键PK参数计算
# 设置PK参数计算的单位转换表ppunits <- pknca_units_table(concu="ug/mL", doseu="mg/kg", amountu="mg", timeu="hr")conc_raw <- arrange(pc, SUBJID,ATPTN)# 设置浓度数据conc_obj_multi <- PKNCAconc(data=conc_raw,formula=AVAL~ATPTN|SUBJID)# 设置剂量数据dose_raw <- select(conc_raw,ATPTN,DOSE,SUBJID) %>% distinct()dose_obj_multi <- PKNCAdose(dose_raw,DOSE~ATPTN|SUBJID,route="intravascular",duration=1.5)# 设置参数计算时间区间intervals_manual <- data.frame(start=0,end=504,cmax=TRUE,auclast=TRUE,aucinf.obs=TRUE)o_data <- PKNCAdata(conc_obj_multi, dose_obj_multi,intervals=intervals_manual,units=ppunits)# 计算PK参数o_results <- pk.nca(o_data)# 结果呈现ncasingle <- o_results[["result"]] %>%select(SUBJID,start,end,PPTESTCD,PPORRES,PPORRESU) %>%filter(PPTESTCD %in% c("cmax","auclast","aucinf.obs"))# 输出PK参数kable(pivot_wider(ncasingle,id_cols=1,names_from = "PPTESTCD", values_from = "PPORRES"))
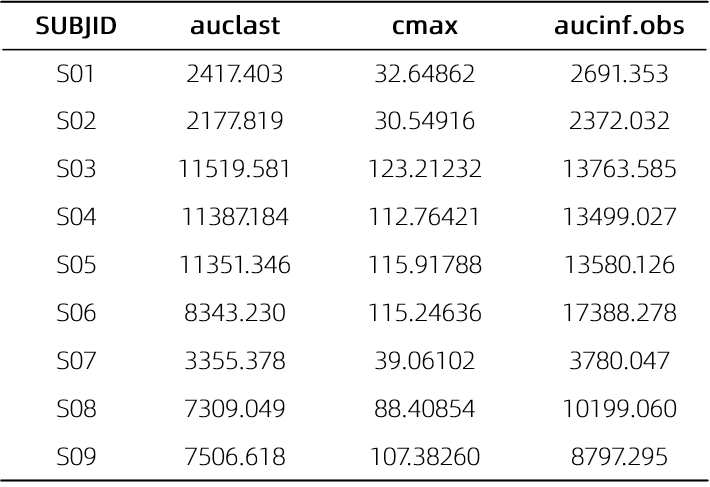

WinNonlin计算结果
PART 03
Power Model分析

剂量-暴露量关系建模
Power Model (原始形式):
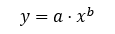
线性化形式 (取自然对数后):
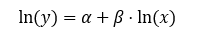
·
数据转换:对剂量和PK参数取自然对数,转化为线性模型。
·
拟合与检验:使用lm函数进行回归分析,计算斜率置信区间和R²,评估模型拟合优度。
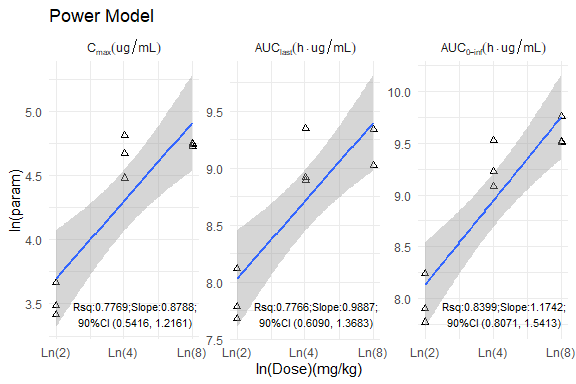
· 可视化:展示不同参数的剂量效应趋势,图中标注斜率、R²及90%置信区间,直观支持统计结论。
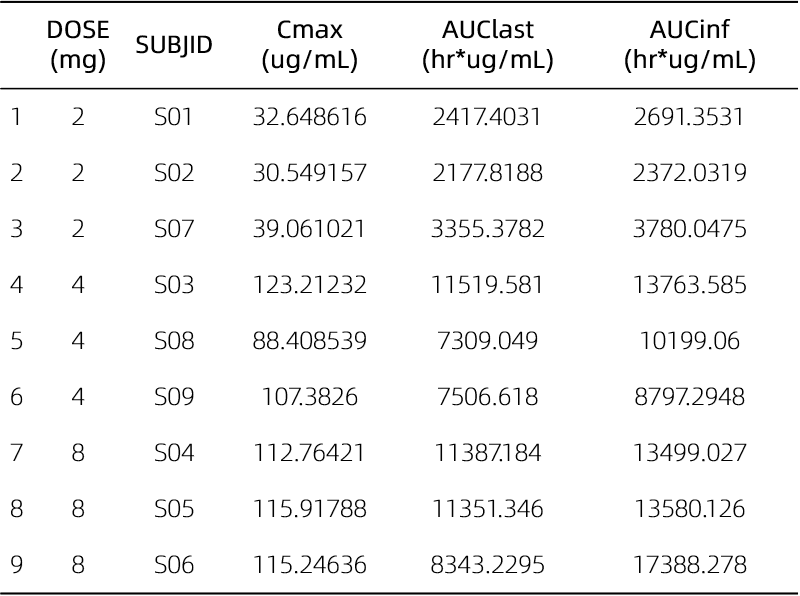
# 剂量与参数的自然对数rst <- left_join(ncasingle,distinct(dose_raw,DOSE,SUBJID),by="SUBJID") %>%mutate(logx=log(DOSE),logy=log(PPORRES),PPTESTCD=factor(PPTESTCD,levels=c("cmax","auclast","aucinf.obs")))# 模型拟合split <- split(rst,~PPTESTCD,drop=TRUE)lmci <- map(split,\(x) data.frame(PPTESTCD=unique(x$PPTESTCD),logx=lm(logy~logx,x)[["coefficients"]][["logx"]],Rsq=summary(lm(logy~logx,x))[["r.squared"]],confint(lm(logy~logx,x), "logx", level= 0.90 ))) %>%bind_rows() %>%# 设置绘图呈现格式mutate(.logx=formatC(`logx`,digits=4,format = "f"),lwci=formatC(`X5..`,digits=4,format = "f"),upci=formatC(`X95..`,digits=4,format = "f"),Rsq=formatC(`Rsq`,digits=4,format = "f"),ci=str_c("Rsq:",Rsq,";Slope:",.logx,";\n90%CI (",lwci,", ",upci,")"),) %>%select(PPTESTCD,ci)# 剂量线性绘图呈现(base <- ggplot(rst,aes(x=logx,y=logy)) +geom_point(shape=24) +geom_smooth(method = "lm", se = TRUE) +geom_text(aes(x=1.5,y=-Inf,label=ci),data=lmci,size=3,vjust=-0.5) +scale_x_continuous(breaks=c(0.69,1.38,2.08),labels=c("Ln(2)","Ln(4)","Ln(8)")) +labs(title = "Power Model",x="ln(Dose)(mg/kg)",y="ln(param)") +facet_wrap(vars(PPTESTCD),scales = "free", axes="all" ,labeller = as_labeller(c("cmax"="C[max](ug/mL)","auclast"="AUC[last](h%.%ug/mL)","aucinf.obs"="AUC[0-inf](h%.%ug/mL)"),,default = label_parsed)) +theme_minimal())

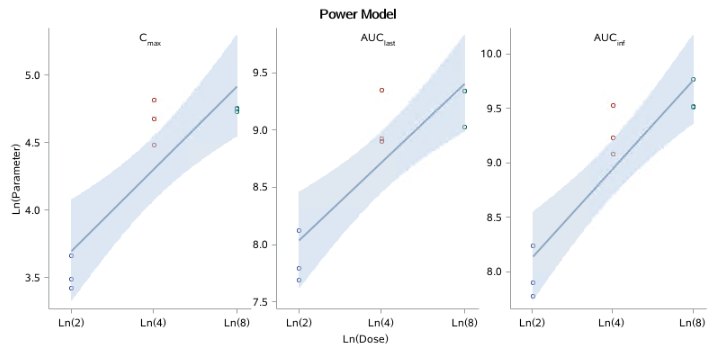
SAS 结果
通过SAS代码复现分析,结果一致性验证了R流程的可靠性。
PART 04
可视化分析

平均血药浓度曲线
·
线性尺度:展示各剂量组浓度随时间的变化趋势,误差条表示标准差,反映个体间变异。
· 对数尺度:通过scale_y_log10转换,突出低浓度阶段的细节,适用于宽动态范围的数据。
pcsas % mutate( aval=as.numeric(AVAL), DOSE=str_c(DOSE,DOSEU) ) # 分组时点血药浓度定量统计stplot % group_by(DOSE,ATPTN) %>% summarise( Mean=mean(aval,na.rm = TRUE), SD=sd(aval,na.rm = TRUE), lower=ifelse(is.na(SD) | Mean-SD % ungroup()# 线性血药浓度-时间曲线(normal % ggplot(aes(x = ATPTN, y = Mean ,colour=DOSE)) + geom_errorbar(mapping=aes(ymin=lower, ymax=upper),width = 0.5,alpha=0.5) + geom_line(linewidth = 0.5,alpha=0.5) + geom_point(alpha=0.5) + scale_x_continuous(breaks=c(24,72,168,336,504)) + labs(x="Time(h)",y="Concentration(mean)",colour="剂量组:",title = "药时曲线") + theme_classic() )
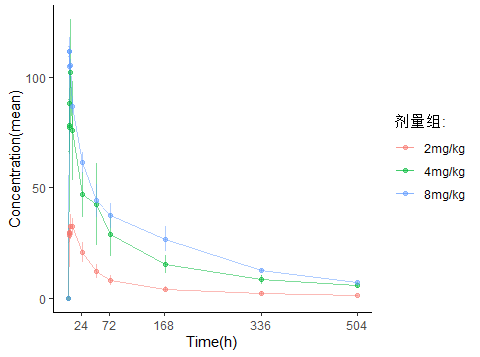
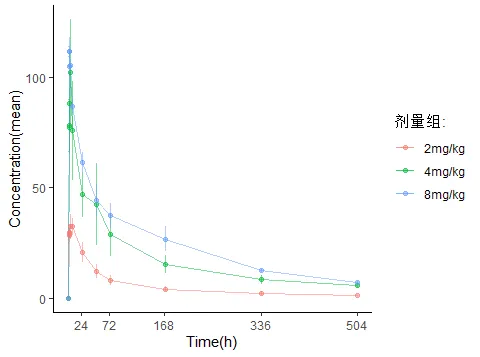
图 药时曲线
# 半对数血药浓度-时间曲线normal %+% subset(stplot, Mean>0) + scale_y_log10(n.breaks=4,labels = scales::trans_format("log10", scales::math_format(10^.x))) + annotation_logticks(sides = "l",outside = TRUE) + expand_limits(y = c(1,10,100)) + coord_cartesian(clip = "off")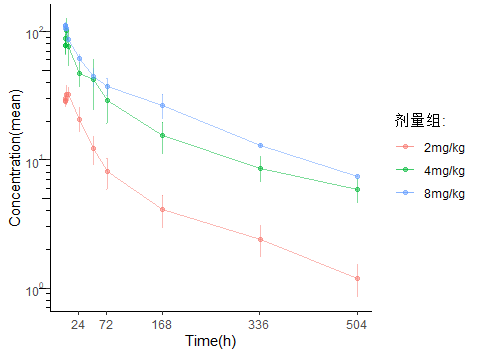
图 药时曲线
PART 05
报告生成

Word文档自动导出
工具链:r2rtf包支持从数据汇总到RTF格式报告的全流程自动化,功能类似SAS的Proc Report。
关键步骤:
·
数据汇总:按剂量组和时间点计算均值、标准差、几何均值等统计量。
·
表格格式化:通过pivot_wider和arsenal包生成结构化表格。
·
RTF输出:定制页面方向(横向)、边距和分栏布局,生成格式精美、便于审阅的标准化报告。
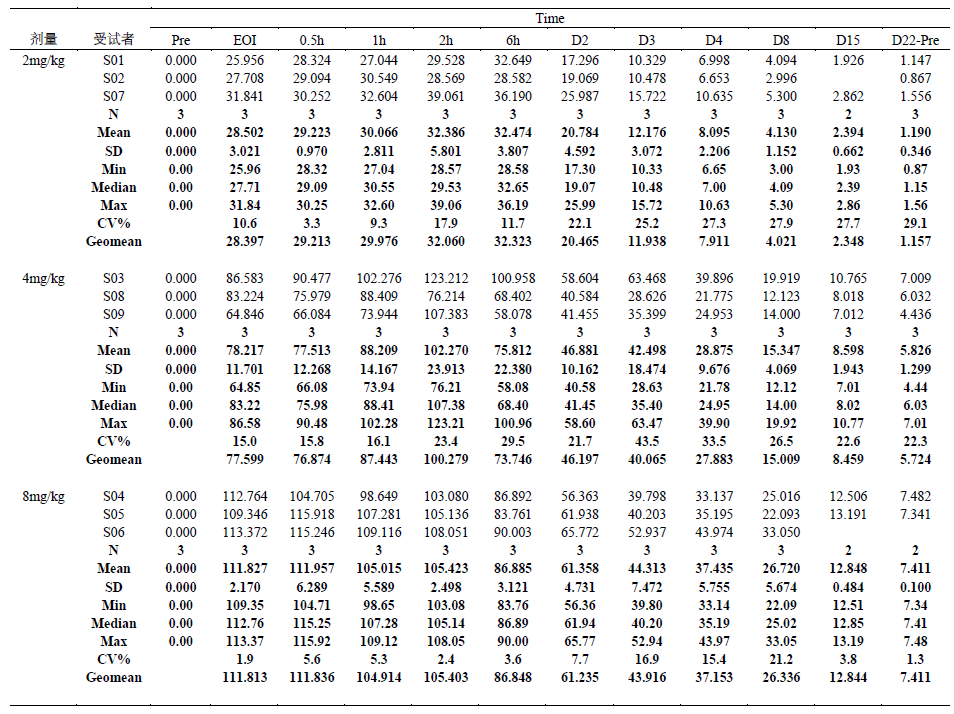
pcsas <- pc %>%mutate(aval=as.numeric(AVAL),log=ifelse(aval>0,log(aval),NA),order=1,DOSE=str_c(DOSE,DOSEU),PCSTRESC=str_trim(formatC(AVAL,digits = 3,format = "f")) ,)# 定量统计,并调整样式st <- filter(pcsas,!is.na(aval)) %>%group_by(DOSE,ATPTN) %>%summarise(N=paste(sum(!is.na(aval))),Mean=str_trim(formatC(mean(aval,na.rm = TRUE),digits = 3,format = "f")),SD=str_trim(formatC(sd(aval,na.rm = TRUE),digits = 3,format = "f")) ,Min=formatC(min(aval,na.rm = TRUE),digits = 2,format = "f"),Median=str_trim(formatC(median(aval,na.rm = TRUE),digits = 2,format = "f")),Max=formatC(max(aval,na.rm = TRUE),digits = 2,format = "f"),`CV%`=case_when(mean(aval,na.rm = TRUE) > 0 ~ formatC(sd(aval,na.rm = TRUE)/mean(aval,na.rm = TRUE)*100,digits = 1,format = "f"),.default = NA),Geomean=case_when(mean(aval,na.rm = TRUE) > 0 ~ str_trim(formatC(exp(mean(log,na.rm = TRUE)),digits = 3,format = "f")),.default = NA),) %>%pivot_longer(cols = c("N","Mean","SD","Min","Median","Max","CV%","Geomean"),names_to = "SUBJID",values_to = "PCSTRESC") %>%mutate(order=case_when(SUBJID=="N" ~ 2 ,SUBJID=="Mean" ~ 3 ,SUBJID=="SD" ~ 4 ,SUBJID=="Min" ~ 5 ,SUBJID=="Median" ~ 6 ,SUBJID=="Max" ~ 7 ,SUBJID=="CV%" ~ 8 ,SUBJID=="Geomean" ~ 9),SUBJID=str_c("\\b1{",SUBJID,"}"),PCSTRESC=str_c("\\b{",PCSTRESC,"}"))# 合并原始数据与统计结果rst <- bind_rows(pcsas,st) %>%select(DOSE,order,SUBJID,ATPTN,PCSTRESC) %>%arrange(DOSE,order,SUBJID,ATPTN) %>%pivot_wider(id_cols = c(1:3),names_from = c("ATPTN"),values_from = "PCSTRESC") %>%ungroup() %>%select(-order) %>%group_by(DOSE) %>%group_modify(~ add_row(.x))# 设置输出样式rtf <- rst %>%rtf_page(orientation = "landscape",border_first = "single",border_last = "single",margin = c(0.75, 0.75, 0.75, 0.75, 0.5, 0.5),nrow=100) %>%rtf_colheader("||Time",border_left=NULL,border_right = NULL,border_top = "single",border_bottom = c("","","single"),col_rel_width = c(8,8,96)) %>%rtf_colheader(colheader = "剂量|受试者|Pre|EOI|0.5h|1h|2h|6h|D2|D3|D4|D8|D15|D22-Pre",border_left=NULL,border_right = NULL,border_top = NULL,border_bottom = "single",,col_rel_width = rep(9,14)) %>%rtf_body(border_left = NULL,border_right = NULL,pageby_row = "column",group_by="DOSE",subline_by=NULL) %>%rtf_encode() %>%str_replace('fcharset161','fcharset134') %>%write_rtf(str_c("outputs/","table_pc_summary.rtf"))
表 模拟数据结果
PART 06
总结
本文通过R语言实现了药代动力学的全流程分析,涵盖数据预处理、参数计算、统计建模、可视化及报告生成,适用于非递交的PK数据分析。熙宁生物凭借专业的PK编程团队和资深药理专家,结合验证工具(如Winnonlin、SAS)与开源软件(如R),能够高效精准地满足客户各类PK/PD统计分析需求,为药物研发与临床研究提供可靠支持。
PART 07
熙宁生物临床药理服务平台
熙宁生物临床药理服务平台,专注于创新药(涵盖生物大分子及小分子)的临床药理学研究,拥有丰富的项目经验和专业的技术能力。我们提供全方位的临床药理学服务,包括:
➢
PK NCA分析:采用WinNonlin软件非房室模型(NCA)计算PK参数,结合SAS和R等专业软件进行数据编程,确保分析结果精准可靠。
➢
实时分析支持剂量爬坡:通过实时数据分析,为SMC会议提供科学依据,优化临床试验设计。
➢
临床药理学研究设计与方案撰写:基于客户需求,定制化设计研究方案,确保科学性和合规性。
➢
完整PK/PD统计分析及CSR撰写:提供生物分析检测到统计分析报告生成的一站式服务,确保高效交付。
平台配备资深的临床药理专家、专业统计师及统计编程团队,能够对统计分析报告中的数据进行深度解读,提供专业的洞见和建议。我们严格按照CDISC标准进行统计编程,确保交付成果符合NMPA、FDA等国际监管机构的要求,助力药物快速通过审批。
PART 08
我们的优势
● 在生物大分子领域,我们拥有丰富的PK统计分析经验,涵盖 CAR-T细胞疗法、单抗、双抗、ADC(抗体药物偶联物)等药物类型,能够精准分析其非线性动力学特征、靶点介导的药物处置(TMDD)等特殊机制。
● 对小分子药物,我们深入分析其吸收、分布、代谢和排泄(ADME)特性,全面评估其药代动力学行为。
熟练使用 SAS 和 R 进行数据编程,生成高度定制化的高质量图表,相比 WinNonlin 的基础功能,R 和 SAS 在灵活性和可视化效果上更具优势,确保数据呈现清晰直观。
凭借高效的团队协作和先进的技术工具,我们能够快速响应客户需求,提供高效、精准的交付成果,助力药物研发加速推进。
参考文献:
[1] The PKNCA R Package
[2] Introduction to r2rtf
[3] Phoenix WinNonlin User’s Guide